Protocole pour la Visualisation des Biofilms Bactériens et la Préparation des Échantillons pour la Microscopie Électronique à Balayage
Protocole pour les Nouveaux Utilisateurs
Protocole pour la visualisation des biofilms bactériens et la préparation d'échantillons pour la microscopie électronique à balayage
Ce protocole utilisateur décrit une méthode pour faire croître un biofilm bactérien dans la µ-Slide I Luer 0.8 (ibidi, 80196) dans des conditions statiques, suivi de sa visualisation à l'aide de la microscopie confocale à balayage laser (CSLM) et de la microscopie électronique à balayage (SEM). Pseudomonas aeruginosa DSM 50071T a été ensemencé dans la µ-Slide I Luer 0.8, puis laissé croître pendant trois jours. Pour favoriser l'attachement cellulaire et la formation de biofilms à la surface, le milieu de culture a été renouvelé après environ 36 heures d'incubation, ce qui a permis d'éliminer les cellules planctoniques. Pour confirmer la formation du biofilm, le milieu de culture a été complété avec un traceur optique, EbbaBiolight 680. Cette molécule est non toxique et donc compatible avec l'imagerie en direct. Elle émet une fluorescence rouge lorsqu'elle se lie aux protéines et aux polysaccharides des matrices extracellulaires, constituant ainsi un marqueur visuel pour les biofilms bactériens. Sur la base de la coloration de l'ADN avec le colorant DAPI et de l'imagerie à l'aide de la CLSM, les cellules bactériennes se sont attachées et ont principalement formé une mono- ou une bilame. Les structures de biofilm plus épaisses ont pu être identifiées par une augmentation du signal de fluorescence émis par le traceur optique. L'objectif était de combiner plusieurs techniques d'imagerie sur la même lame. Ainsi, après l'imagerie CLSM, les cellules ont été fixées sur la même lame, déshydratées, puis séchées pour l'imagerie SEM. Un moule en 3D a été créé en interne pour isoler soigneusement la lamelle de couverture, évitant ainsi de perturber l'organisation spatiale des cellules attachées. La couche cellulaire bactérienne formée a ensuite été imagée à des grossissements allant de 2 000x à 15 000x sur la lamelle de couverture isolée.
Documents Connexes
• Instructions pour la µ-Slide I Luer (PDF)
• Protocole III : Suivi des biofilms dans les cultures vivantes en utilisant EbbaBiolight 680
Related Documents
• Instructions µ-Slide I Luer (PDF)
• Protocol III: Biofilm tracking in live cultures using EbbaBiolight 680
1 Matériaux et Réactifs
1.1 Cellules et Réactifs
• Culture de Pseudomonas aeruginosa (DSMZ, 50071)
• Tryptone (Thermo Fisher Scientific, 211705)
• Peptone de farine de soja (Millipore, 1.07212)
• Glucose (VWR, 24379)
• Chlorure de sodium (NaCl, VWR, 27800)
• Phosphate dipotassique (K2HPO4, Sigma, P3786)
• Tampon phosphate salin (PBS, Sigma, P7994)
• EbbaBiolight 680 (EbbaBiotech AB)
• 4',6-Diamidino-2-Phénylindole (DAPI, concentration stock 1 mg/mL, Thermo Fisher Scientific, 62248)
• Glutaraldéhyde à 25% (Sigma, G7776)
Protocole Utilisateur 14
• Eau déionisée (diH2O)
• Éthanol absolu (Sigma, 34852-M)
1.2 Tampons et Solutions
Milieu de Culture
• Bouillon de soja tryptique (TSB ; 17 g/L de tryptone, 3 g/L de peptone de farine de soja, 2,5 g/L de glucose, 5 g/L de NaCl et 2,5 g/L de K2HPO4)
• TSB supplémenté avec EbbaBiolight 680 en proportion 1:1000 Tampon de Lavage
• PBS 1x
Solution de Coloration
• DAPI en proportion 1:1000 dans PBS 1x (concentration finale 1 µg/mL)
Dilutions d'Éthanol Absolu à 30%, 50%, 70%, 90%, 100% (v/v)
• Mélanger les volumes appropriés de diH2O et d'éthanol absolu
1.3 Équipement
• µ-Slide I Luer 0.8, ibiTreat (ibidi, 80196)
• Hotte à flux laminaire
• Hotte aspirante
• Incubateur, 30°C
• Incubateur, 55°C
• Pipettes
• Réfrigérateur
• Dessiccateur
• Scalpel ou lame
• Ciseaux
• Pincettes
• Moule de découpe (voir exemple dans la Figure 1)
• Microscope confocal inversé (ici : ZEISS LSM 880)
• ZEN black 2.3 SP1
• (Fiji Is Just) ImageJ 1.54f
• Microscope électronique à balayage (ici : JEOL JSM-6480LV)
• Système de dorure (ici : JEOL JFC-1200 fine coater)
• Ruban adhésif en carbone
• JEOL JSM-6480 version 7.07
| Figure 1: Schéma du moule de découpe utilisé pour découper la lamelle de couverture de la µ-Slide I Luer 0.8. Les lignes blanches représentent l'emplacement de la découpe, le long de la µ-Slide (en bas) et juste à l'extérieur des réservoirs (en haut). |
|
2 Procédure
2.1 Attachement du Biofilm Bactérien
Veuillez lire les Instructions avant de travailler avec la µ-Slide I Luer 0.8 et suivre les recommandations pour le pipetage général et le changement de solutions dans la µ-Slide. Effectuez toutes les étapes dans des conditions stériles. Avant de commencer l'expérience, préparez Pseudomonas aeruginosa DSM 50071 en tant que culture de nuit en inoculant la souche dans un milieu LB dans un flacon Erlenmeyer (par exemple, 100 mL) à 30°C et 160 rpm dans l'obscurité. Diluez la culture 1:100 dans le milieu contenant l'optotraceur 1:1000, selon les instructions du fournisseur. Notez que la concentration cellulaire n'a pas été ajustée aux valeurs recommandées par le fournisseur.
Note Importante: Équilibrez les matériaux requis, tels que les µ-Slides, pendant la nuit à l'intérieur de l'incubateur à 30°C. L'équilibrage est essentiel pour éviter l'apparition de bulles d'air au fil du temps.
- Injectez 200 µL de cellules diluées dans le canal de la µ-Slide I Luer 0.8 et fermez avec les capuchons fournis selon les Instructions.
- Laissez les cellules se déposer et s'attacher pendant 2 heures à température ambiante (RT) dans l'obscurité.
- Ajoutez 60 µL de milieu TSB supplémenté avec l'optotraceur dans chaque réservoir et couvrez-les avec les capuchons.
- Incubez la µ-Slide à 30°C dans l'obscurité et dans des conditions statiques pendant 1 à 2 jours.
- Échangez le milieu en prélevant 100 µL d'un des réservoirs et en ajoutant 100 µL de TSB fraîchement préparé supplémenté avec l'optotraceur, selon les Instructions.
- Répétez l'étape précédente 5 fois à 10 fois pour assurer une substitution complète du milieu, en évitant les bulles d'air.
- Remplissez les réservoirs de la µ-Slide avec 60 µL de milieu TSB supplémenté sans cellules.
- Incubez la µ-Slide à 30°C dans l'obscurité et dans des conditions statiques pendant 1 à 2 jours pour favoriser davantage l'attachement et le développement du biofilm.
2.2 Coloration DAPI et Observation Sous la CLSM
La coloration est réalisée directement dans la µ-Slide I Luer 0.8. Effectuez toutes les étapes sous la hotte aspirante.
Note Importante: Évitez de retirer le volume total du canal en une seule fois. Les cellules ne doivent pas sécher.
- Pour remplacer le milieu par du PBS 1x, commencez par retirer 100 µL de milieu d'un réservoir. Ensuite, ajoutez 100 µL de solution de lavage PBS 1x à l'autre réservoir.
- Répétez ce processus entre 5 et 10 fois, jusqu'à ce que le milieu ait été complètement remplacé par du PBS 1x.
- Remplacez le PBS 1x par la solution de coloration DAPI, en suivant la technique de pipetage décrite précédemment. Assurez-vous que le canal est complètement rempli de la solution de coloration DAPI.
- Incubez la µ-Slide pendant 10 minutes à température ambiante dans l'obscurité et dans des conditions statiques.
- Lavez l'excès de colorant avec du diH2O deux fois, en suivant la technique de pipetage décrite précédemment.
- Effectuez un dernier lavage avec du PBS 1x, en suivant la technique de pipetage décrite précédemment.
- Imager les cellules attachées en utilisant une CLSM (Figure 2).
- Conservez la µ-Slide dans le réfrigérateur à 4°C (pendant un maximum de deux jours sauf si l'échantillon est fixé) jusqu'à un traitement ultérieur.
 |
Figure 2 : Microscopie confocale d'un biofilm de P. aeruginosa coloré (rouge : EbbaBiolight 680 (Cy3.5), bleu : DAPI). Objectif 63x. Les cellules sont principalement attachées en monocouche. Certaines structures tridimensionnelles ressemblant à un biofilm sont visibles, notamment dans les sections où le signal rouge est plus intense, mettant en évidence la liaison de l'optotraceur aux composants du biofilm. |
2.3 Préparation des échantillons pour l'observation au SEM
La préparation des échantillons est réalisée directement dans la µ-Slide I Luer 0.8. Effectuez toutes les étapes sous la hotte aspirante.
Note Importante: Évitez de retirer le volume total du canal en une seule fois. Les cellules ne doivent pas sécher. Toutes les étapes de lavage et d'échange de solutions ont été effectuées conformément aux Instructions, en retirant 100 µL de solution 1 d'un des réservoirs et en ajoutant 100 µL de solution 2 au réservoir opposé. Ce processus a été répété minutieusement pour assurer l'échange complet des solutions.
- Fixez les cellules attachées en remplaçant le PBS 1x dans le canal et les réservoirs par du glutaraldéhyde à 2,5% dans du PBS 1x, en suivant la technique de pipetage décrite à l'étape 2.2, première étape.
- Incubez la µ-Slide pendant 3 à 4 heures à température ambiante (ou toute la nuit à 4°C).
- Lavez les cellules au moins 5 fois avec du PBS 1x pour éliminer complètement la solution de glutaraldéhyde, en suivant la technique de pipetage décrite précédemment.
- Déshydratez séquentiellement les biofilms attachés en les incubant dans des dilutions ascendantes d'éthanol absolu à 30%, 50%, 70% et 90% pendant 15 minutes chacune.
- Déshydratez les cellules dans de l'éthanol à 100% deux fois pendant 20 minutes chacune.
- Laissez le canal sécher dans l'incubateur à 55°C sans les capuchons jusqu'à ce que le liquide soit complètement évaporé (ici, pendant 2 heures).
- Conservez la µ-Slide dans le dessiccateur jusqu'au traitement ultérieur.
- Découpez la lamelle de couverture avec un scalpel ou une lame, selon le diagramme (Figure 3).
- Gardez la lamelle de couverture découpée debout dans le dessiccateur jusqu'au traitement ultérieur (pendant plusieurs semaines).
- Découpez la lamelle de couverture en segments plus petits (environ 1 cm de long) et recouvrez-la deux fois d'or (Figure 4) avant de l'observer sous le microscope électronique.

|
Figure 3: Diagramme de la lamelle de couverture découpée de la µ-Slide I Luer 0.8. Les lignes rouges en pointillés représentent où les coupes sont réalisées, suivant le moule, et la section colorée représente la lamelle de couverture qui peut être soigneusement retirée après la découpe. |

|
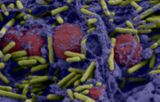
Figure 3: Microscopie électronique à balayage des cellules de P. aeruginosa et du biofilm attaché à la lamelle de couverture de la µ-Slide I Luer 0.8 visualisé avec (A) un grossissement de 2 000x et (B) un grossissement de 7 500x. |
Ce protocole utilisateur est un protocole évalué par les pairs d'ibidi provenant d'un utilisateur réel. ibidi ne garantit ni sa fonctionnalité ni sa reproductibilité. Pour ce protocole utilisateur, ibidi ne fournit qu'un soutien limité. Veuillez contacter l'auteur correspondant pour des informations détaillées. À usage de recherche uniquement.


